2019年1月份,经过严格的同行匿名评审,国际生物信息权威杂志Bioinformatics(影响因子5.48)在线发表了中科普瑞自主研发的融合基因算法软件BreakID,BreakID是根据靶向二代测序数据中识别融合事件并准确鉴定融合断点位置的软件。对比其它融合鉴定软件,BreakID具有较高的灵敏度和特异性,运算速度快,占用内存小,对于临床检测更加准确,快速。

文章链接:
https://academic.oup.com/bioinformatics/advance-article-abstract/doi/10.1093/bioinformatics/bty1070/5270666
如下图所示,BreakID算法涵盖3个步骤:
1. 扫描肿瘤样本测序比对数据,识别出不正常比对读长对(Discordant Read Pairs),并应用合成聚类合并算法(Agglomerative Hierarchical Clustering, AHC)对其进行聚类;
2. 识别经过断点的断裂读长(Split Reads)得到断点的具体基因组位置,推断出候选融合事件;
3. 对融合事件进行过滤(去除假阳性)、分类及相关临床信息注释。
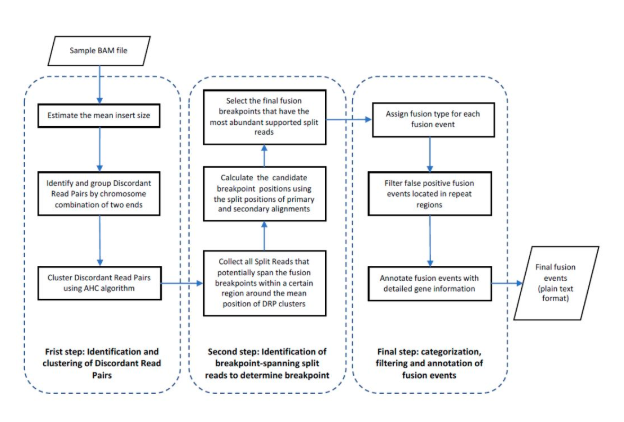
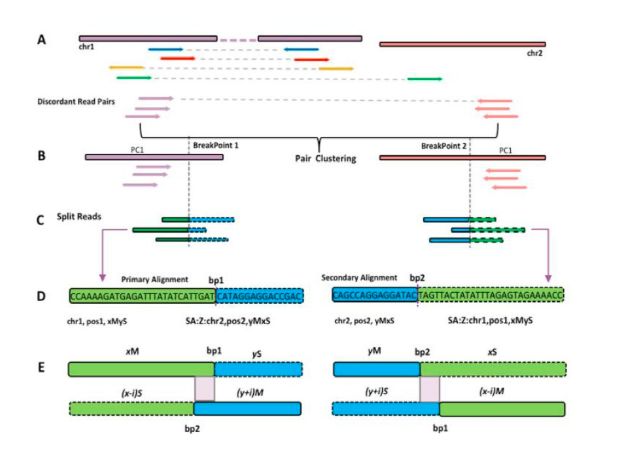
BreakID基因融合断点的分析模型:
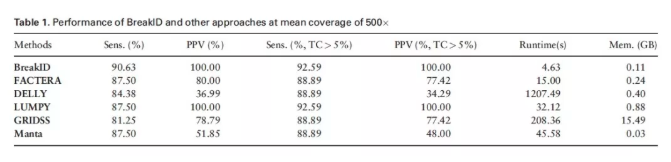
文章中比较了6种不同软件对11个验证样本的融合基因检测结果,发现我司自主研发的BreakID软件在测序深度达到500X,其检测灵敏度达到90.63%,特异性达到100%,显著优于其他算法,并且在运行速度及内存占用方面较其他软件有了较大的提高。
凭借自主研发的一系列生信分析算法Sinotools(点突变、小片段插入缺失、拷贝数变异及基因融合),我司通过了2018年上海市临床检验中心开展的肿瘤体细胞基因突变高通量测序检测生物信息分析室间质量评价, 并且在基因融合突变方面尤为突出。

中科普瑞肿瘤基因检测服务中的基因融合检测基于BreakID算法,结合大规模样本验证、优化,全面应用于非小细胞肺癌患者的ALK、ROS1、RET融合,泛癌种NTRK的融合突变以及其他重要融合的用药、耐药检测等,为患者和临床医生找到准确靠谱的用药指导方案。
技术的进步离不开自己本身的不懈探索以及行业学者的专业认可,BreakID 软件源代码可以在github网站上免费获得(https://github.com/SinOncology/BreakID),中科普瑞热切欢迎能和行业精英在肿瘤精准用药领域共同探索交流,加深对自身领域的理解和认识,持续进步、不断迭代,持续鞭策自身,开发出更准确,更完美的肿瘤精准用药检测产品。中科普瑞的宗旨是:“普及精准医疗,瑞耀生命之光——让中国没有难治的癌症”。
附百度网盘全文下载链接:
https://pan.baidu.com/s/1LVY-931SjM2MsNEAM_KSGA


